c:ms:2025:w07_anova_note
This is an old revision of the document!
Table of Contents
ANOVA in R
#
# ANOVA test with 4 levels in IV
#
rm(list=ls())
set.seed(101)
rnorm2 <- function(n,mean,sd){ mean+sd*scale(rnorm(n)) }
n <- 30
na <- n
nb <- n
nc <- n
nd <- n
mean.a <- 26
mean.b <- 25
mean.c <- 23
mean.d <- 22
A <- rnorm2(na, mean.a, sqrt(1160/(na-1)))
B <- rnorm2(nb, mean.b, sqrt(1000/(nb-1)))
C <- rnorm2(nc, mean.c, sqrt(1000/(nc-1)))
D <- rnorm2(nd, mean.d, sqrt(1000/(nd-1)))
# A combined group with group A and B
# We call it group total
# we can obtain its mean, variance, ss, df, etc.
#
A
B
C
D
comb <- data.frame(A, B, C, D)
dat <- stack(comb)
head(dat)
colnames(dat)[1] <- "values"
colnames(dat)[2] <- "group"
head(dat)
mean.total <- mean(dat$values)
var.total <- var(dat$values)
# variance를 ms라고 부르기도 한다
ms.total <- var.total
df.total <- length(dat$values) - 1
ss.total <- var.total * df.total
ss.total.check <- sum(
(dat$values - mean(dat$values))^2
)
mean.total
var.total
ms.total
df.total
ss.total
ss.total.check
# Now for each group
mean.a <- mean(A)
mean.b <- mean(B)
mean.c <- mean(C)
mean.d <- mean(D)
mean.a
mean.b
mean.c
mean.d
# 그룹 간의 차이에서 나타나는 분산
# 수업시간에 설명을 잘 들을 것
hist(dat$values)
abline(v = mean(dat$values), lty=2, lwd=3, col="red")
abline(v = mean.a, lty=2, lwd=3, col="blue")
abline(v = mean.b, lty=2, lwd=3, col="darkgreen")
abline(v = mean.c, lty=2, lwd=3, col="black")
abline(v = mean.d, lty=2, lwd=3, col="orange")
# mean.total 에서 그룹a의 평균까지의 차이를 구한 후
# 이를 제곱하여 그룹 A 멤버의 숫자만큼 더한다 =
# 즉, SS를 구하는 방법.
# 전체평균에서 그룹평균을 뺀 것의 제곱을
# 그룹 구성원 숫자만큼 더하는 것
# 그리고 이들을 다시 모두 더하여
# ss.between에 저장
length(A) * ((mean.total - mean.a)^2)
length(B) * ((mean.total - mean.b)^2)
length(C) * ((mean.total - mean.c)^2)
length(D) * ((mean.total - mean.d)^2)
ss.between <-
length(A) * ((mean.total - mean.a)^2) +
length(B) * ((mean.total - mean.b)^2) +
length(C) * ((mean.total - mean.c)^2) +
length(D) * ((mean.total - mean.d)^2)
ss.between
# df between group은 연구에 사용된
# 그룹의 숫자에서 1을 뺀 숫자
n.groups <- nlevels(dat$group)
df.between <- n.groups - 1
# 이 그룹 간 차이에 기인하는 분산 값은
ms.between <- ss.between / df.between
# 한편 ss.a 와 ss.b는 각 그룹 내의
# 분산을 알아보기 위한 방법
df.a <- length(A) - 1
df.b <- length(B) - 1
df.c <- length(C) - 1
df.d <- length(D) - 1
ss.a <- var(A) * df.a
ss.b <- var(B) * df.b
ss.c <- var(C) * df.c
ss.d <- var(D) * df.d
ss.within <- ss.a + ss.b + ss.c + ss.d
df.within <- df.a + df.b + df.c + df.d
ms.within <- ss.within / df.within
# 여기까지 우리는
# 전체분산
# 그룹간분산
# 그룹내분산 값을
# 구한 것
# ms.between은 그룹의 차이때문에 생긴
# 분산으로 IV 혹은 treatment 때문에 생기는
# 차이에 기인하는 분산이고
# ms.within은 각 그룹 내부에서 일어나는 분산이므로
# (variation이므로) 연구자의 관심사와는 상관이 없이
# 나타나는 random한 분산이라고 하면
# t test 때와 마찬가지로
# 그룹의 차이 / 랜덤 차이를 (에러 -> 분산은 에러라고도 했다)
# 구해볼 수 있다.
# 즉, 그룹갑분산은 사실 = diff (between groups)
# 그리고 그룹내 분산은 사실 = re
# 따라서 우리는 위 둘 간의 비율을 t test와 같이
# 살펴볼 수 있다
# 이것을 f.calculated 이라고 하고
f.calculated <- ms.between / ms.within
# 이 값을 출력해 본다
f.calculated
# 컴퓨터 계산이 쉬워지기 전에는 아래처럼 0.5 level
# 에서의 f값을 구한 후 이것과 계산된 f값을 비교해봤었다.
qf(.05, df1 = df.between, df2 = df.within, lower.tail = FALSE)
f.calculated
# 위에서 f.calculated > qf값이므로
# f.calculated 값으로 영가설을 부정하고
# 연구가설을 채택하면 판단이 잘못일 확률이
# 0.05보다 작다는 것을 안다.
# 그러나 컴퓨터계산이 용이해지고서는 qf대신에
# pf를 써서 f.cal 값에 해당하는 prob. level을
# 알아본다.
# percentage of f distribution with
# df1 and df2 option
# 이는 그림의 왼쪽을 나타내므로
# 차이가 점점 커지게 되는 오른쪽을
# 계산하기 위해서는 1-x를 취한다
f.calculated.pvalue <- 1-pf(f.calculated, df1=df.between, df2=df.within)
f.calculated.pvalue
f.calculated
# graph 로 이해
x <- rf(500000, df1 = df.between, df2 = df.within)
y.max <- max(df(x,df1=df.between, df2=df.within))
hist(x,
breaks = "Scott",
freq = FALSE,
xlim = c(0, f.calculated + 1),
ylim = c(0, y.max + .3),
xlab = "",
main = paste("Histogram for
a F-distribution with
df1 = ", df.between,
"and df2 = ", df.within,
"F calculated value = ", round(f.calculated,3),
"p value = ", f.calculated.pvalue),
cex.main = 0.9
)
curve(df(x, df1 = df.between, df2 = df.within),
from = 0, to = f.calculated + 1, n = 5000,
col = "red", lwd = 2,
add = T)
abline(v=f.calculated, col="blue", lwd=2, lty="dotted")
f.calculated
f.calculated.pvalue
1 - f.calculated.pvalue
# Now check this
ss.total
ss.between
ss.within
ss.total
ss.between + ss.within
# 한편 df는
# df.total 30 - 1
df.total
df.between
df.within
df.total
df.between + df.within
##################################################
a.res <- aov(values ~ group, data=dat)
a.res.sum <- summary(a.res)
a.res.sum
# 그러나 정확히 어떤 그룹에서 차이가 나는지는 판단해주지 않음 pairwise.t.test(dat$values, dat$group, p.adj = "none") # OR pairwise.t.test(dat$values, dat$group, p.adj = "bonf") pairwise.t.test(dat$values, dat$group, p.adj = "holm") # OR TukeyHSD(anova.output) TukeyHSD(a.res) boxplot(dat$values~dat$group) f.calculated f.calculated.pvalue
ANOVA in R: Output
> #
> # 3 샘플 종류 추출
> # A, B, C 학년에 따라서 욕하는 정도가 달라질것이라는
> # 가설
> set.seed(201)
> rnorm2 <- function(n,mean,sd){ mean+sd*scale(rnorm(n)) }
> A <- rnorm2(16, 26, sqrt(600/15))
> B <- rnorm2(16, 24, sqrt(750/15))
> C <- rnorm2(16, 19, sqrt(900/15))
>
> A <- c(A)
> B <- c(B)
> C <- c(C)
>
> # 평균구하기
> mean(A)
[1] 26
> mean(B)
[1] 24
> mean(C)
[1] 19
>
> # 3 샘플을 합치기
> # 두번재 컬럼 = group A, B, C 가 되도록
> comb3 <- stack(list(a=A, b=B, c=C))
> comb3
values ind
1 28.956987 a
2 24.588298 a
3 29.232040 a
4 15.221730 a
5 18.069720 a
6 26.455426 a
7 28.824505 a
8 27.362726 a
9 11.013442 a
10 28.284603 a
11 31.778215 a
12 28.525725 a
13 29.735641 a
14 33.996331 a
15 22.487061 a
16 31.467550 a
17 26.638215 b
18 25.537955 b
19 30.194374 b
20 22.161849 b
21 23.164369 b
22 17.436099 b
23 34.602104 b
24 12.091427 b
25 36.147829 b
26 21.460025 b
27 30.381254 b
28 24.367170 b
29 17.691419 b
30 26.351577 b
31 11.330276 b
32 24.444055 b
33 10.842903 c
34 20.414861 c
35 11.872108 c
36 29.195858 c
37 26.074787 c
38 18.262033 c
39 18.863621 c
40 25.906536 c
41 4.379624 c
42 2.980549 c
43 20.825446 c
44 22.038722 c
45 24.175933 c
46 25.745030 c
47 23.796968 c
48 18.625020 c
> colnames(comb3)[1] <- "values"
> colnames(comb3)[2] <- "group"
> comb3
values group
1 28.956987 a
2 24.588298 a
3 29.232040 a
4 15.221730 a
5 18.069720 a
6 26.455426 a
7 28.824505 a
8 27.362726 a
9 11.013442 a
10 28.284603 a
11 31.778215 a
12 28.525725 a
13 29.735641 a
14 33.996331 a
15 22.487061 a
16 31.467550 a
17 26.638215 b
18 25.537955 b
19 30.194374 b
20 22.161849 b
21 23.164369 b
22 17.436099 b
23 34.602104 b
24 12.091427 b
25 36.147829 b
26 21.460025 b
27 30.381254 b
28 24.367170 b
29 17.691419 b
30 26.351577 b
31 11.330276 b
32 24.444055 b
33 10.842903 c
34 20.414861 c
35 11.872108 c
36 29.195858 c
37 26.074787 c
38 18.262033 c
39 18.863621 c
40 25.906536 c
41 4.379624 c
42 2.980549 c
43 20.825446 c
44 22.038722 c
45 24.175933 c
46 25.745030 c
47 23.796968 c
48 18.625020 c
>
> # 전체구성원을 하나로 보고 분산값을 구해본다
> # ms.tot = ss.tot / df.tot
> # ss.tot = sum(xi-mean.tot)^2
> # df.tot = N - 1
> # attach(comb3)
> mean.tot <- mean(comb3$values)
> ss.tot <- sum((comb3$values-mean.tot)^2)
> df.tot <- 48-1
> ms.tot <- ss.tot/df.tot
> ss.tot
[1] 2666
> df.tot
[1] 47
> ms.tot
[1] 56.7234
> var(comb3$values)
[1] 56.7234
>
> # A, B, C 평균
> m.a <- mean(A)
> m.b <- mean(B)
> m.c <- mean(C)
> ms.a <- var(A)
> ms.b <- var(B)
> ms.c <- var(C)
> df.a <- 15
> df.b <- 15
> df.c <- 15
> # 사실 우리는 아래 각 그룹의 SS가
> # 600, 750, 900 인것을 알고 있다.
> ss.a <- ms.a*df.a
> ss.b <- ms.b*df.b
> ss.c <- ms.c*df.c
>
> ss.within <- ss.a + ss.b + ss.c
> ss.within <- ss.within
>
> 16*((m.a-mean.tot)^2)
[1] 144
> 16*((m.b-mean.tot)^2)
[1] 16
> 16*((m.c-mean.tot)^2)
[1] 256
> ss.bet <- 16*((m.a-mean.tot)^2) + 16*((m.b-mean.tot)^2) + 16*((m.c-mean.tot)^2)
>
> ss.tot
[1] 2666
> ss.bet
[1] 416
> ss.within
[1] 2250
> ss.bet + ss.within
[1] 2666
>
> df.tot
[1] 47
> df.within <- df.a + df.b + df.c
> df.within
[1] 45
> df.bet <- 3-1
> df.bet
[1] 2
> df.bet + df.within
[1] 47
>
> ms.bet <- ss.bet/df.bet
> ms.within <- ss.within /df.within
> f.calculated <- ms.bet/ms.within
> ms.bet
[1] 208
> ms.within
[1] 50
> f.calculated
[1] 4.16
> # 이 점수에서의 F critical value =
> fp.value <- pf(f.calculated, df1=2, df2=45, lower.tail = F)
> fp.value
[1] 0.02199143
>
> # f.calculated 와 fp.value로 판단
> f.calculated
[1] 4.16
> fp.value
[1] 0.02199143
>
>
> a.res <- aov(values ~ group, data=comb3)
> a.res.sum <- summary(a.res)
> a.res.sum
Df Sum Sq Mean Sq F value Pr(>F)
group 2 416 208 4.16 0.022 *
Residuals 45 2250 50
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> # 위에서
> # ssd라는 function을 만들면
>
> ssd <- function(x) {
+ var(x) * (length(x)-1) }
> ss.a1 <- ssd(A)
> ss.b2 <- ssd(B)
> ss.c1 <- ssd(C)
>
> ss.a == ss.a1
[1] TRUE
> ss.b == ss.b1
[1] TRUE
> ss.c == ss.c1
[1] TRUE
> # 그러나 정확히 어떤 그룹에서 차이가 나는지는 판단해주지 않음
> pairwise.t.test(comb3$values, comb3$group, p.adj = "none")
Pairwise comparisons using t tests with pooled SD
data: comb3$values and comb3$group
a b
b 0.4279 -
c 0.0075 0.0516
P value adjustment method: none
> # OR
> pairwise.t.test(comb3$values, comb3$group, p.adj = "bonf")
Pairwise comparisons using t tests with pooled SD
data: comb3$values and comb3$group
a b
b 1.000 -
c 0.023 0.155
P value adjustment method: bonferroni
> pairwise.t.test(comb3$values, comb3$group, p.adj = "holm")
Pairwise comparisons using t tests with pooled SD
data: comb3$values and comb3$group
a b
b 0.428 -
c 0.023 0.103
P value adjustment method: holm
>
> # OR TukeyHSD(anova.output)
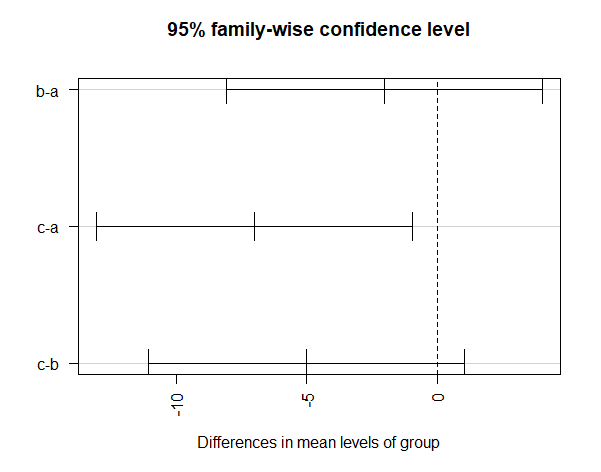
> TukeyHSD(a.res)
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = values ~ group, data = comb3)
$group
diff lwr upr p adj
b-a -2 -8.059034 4.059034 0.7049466
c-a -7 -13.059034 -0.940966 0.0201250
c-b -5 -11.059034 1.059034 0.1238770
>
>
>
Post hoc test
m.a m.b m.c d.ab <- m.a - m.b d.ac <- m.a - m.c d.bc <- m.b - m.c d.ab d.ac d.bc # mse (ms within) from the a.res.sum output # a.res.sum == summary(aov(values ~ group, data=comb3)) a.res.sum # mse = 50 mse <- 50 # 혹은 fansy way from comb3 data.frame # 15 는 각 그룹의 df sse.ch <- sum(tapply(comb3$values, comb3$group, var)*15) sse.ch mse.ch <- sse.ch/45 mse.ch se <- sqrt(mse/length(A)) # now t scores for two compared groups t.ab <- d.ab / se t.ac <- d.ac / se t.bc <- d.bc / se t.ab t.ac t.bc # 이제 위의 점수를 .05 레벨에서 비교할 점수를 찾아 비교한다 # qtukey 펑션을 이용한다 t.crit <- qtukey( .95, nmeans = 3, df = 45) t.crit # t.ac만이 큰 것을 알 수 있다. # 따라서 a 와 c 가 서로 다른 그룹 # 즉, 1학년과 3학년이 서로 다른 그룹 # 혹은 R이 보통 제시한 거꾸로 방법으로 보면 ptukey(t.ab, nmeans=3, df=45, lower.tail = F) ptukey(t.ac, nmeans=3, df=45, lower.tail = F) ptukey(t.bc, nmeans=3, df=45, lower.tail = F) TukeyHSD(a.res, conf.level=.95)
plot(TukeyHSD(a.res, conf.level=.95), las = 2)
pairwise.t.test(comb3$values, comb3$group, p.adj = "bonf")
post hoc test: output
> m.a
[1] 26
> m.b
[1] 24
> m.c
[1] 19
>
> d.ab <- m.a - m.b
> d.ac <- m.a - m.c
> d.bc <- m.b - m.c
>
> d.ab
[1] 2
> d.ac
[1] 7
> d.bc
[1] 5
>
> # se
> # from the a.res.sum output
> a.res.sum
Df Sum Sq Mean Sq F value Pr(>F)
group 2 416 208 4.16 0.022 *
Residuals 45 2250 50
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> # mse = 50
> mse <- 50
>
> se <- sqrt(mse/length(A))
>
> # now t scores for two compared groups
> t.ab <- d.ab / se
> t.ac <- d.ac / se
> t.bc <- d.bc / se
>
> t.ab
[1] 1.131371
> t.ac
[1] 3.959798
> t.bc
[1] 2.828427
>
> # 이제 위의 점수를 .05 레벨에서 비교할 점수를 찾아 비교한다
> # qtukey 펑션을 이용한다
> t.crit <- qtukey( .95, nmeans = 3, df = 45)
> t.crit
[1] 3.427507
>
> # t.ac만이 큰 것을 알 수 있다.
> # 따라서 a 와 c 가 서로 다른 그룹
> # 즉, 1학년과 3학년이 서로 다른 그룹
>
> # 혹은 R이 보통 제시한 거꾸로 방법으로 보면
> ptukey(t.ab, nmeans=3, df=45, lower.tail = F)
[1] 0.7049466
> ptukey(t.ac, nmeans=3, df=45, lower.tail = F)
[1] 0.02012498
> ptukey(t.bc, nmeans=3, df=45, lower.tail = F)
[1] 0.123877
>
> TukeyHSD(a.res, conf.level=.95)
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = values ~ group, data = comb3)
$group
diff lwr upr p adj
b-a -2 -8.059034 4.059034 0.7049466
c-a -7 -13.059034 -0.940966 0.0201250
c-b -5 -11.059034 1.059034 0.1238770
R square or Eta square
SS toal
- = Y 변인만으로 Y를 예측했을 때의 오차의 제곱
- Y 변인만으로 = Y의 평균을 가지고 Y값을 예측한 것
- 학습 초기에 에러의 제곱의 합으로 설명된 것
SS between
- X 변인 (independent variable) 정보가 고려 되었을 때
- 독립변인이 고려되었을 때 (됨으로써)
- 없어지는 SS total의 불확실 성
- 혹은 획득되는 설명력
SS error
- IV가 고려되었음에도 불구하고
- 끝까지 남는 error
SS total = SS between + SS within
SS between / SS total = IV 가 kicked in 되었을 때 없어지는 uncertainty = IV 의 설명력 = R square value
즉, IV로 uncertainty 가 얼마나 없어질까? 라는 아이디어
이를 살펴보기 위해
ss.tot ss.bet r.sq <- ss.bet / ss.tot r.sq # then . . . . lm.res <- lm(values ~ group, data = comb3) summary(lm.res) anova(lm.res)
R square: output
> ss.tot
[1] 2666
> ss.bet
[1] 416
> r.sq <- ss.bet / ss.tot
> r.sq
[1] 0.156039
>
> # then . . . .
>
> lm.res <- lm(values ~ group, data = comb3)
> summary(lm.res)
Call:
lm(formula = values ~ group, data = comb3)
Residuals:
Min 1Q Median 3Q Max
-16.020 -2.783 1.476 4.892 12.148
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 26.000 1.768 14.71 <2e-16 ***
groupb -2.000 2.500 -0.80 0.4279
groupc -7.000 2.500 -2.80 0.0075 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 7.071 on 45 degrees of freedom
Multiple R-squared: 0.156, Adjusted R-squared: 0.1185
F-statistic: 4.16 on 2 and 45 DF, p-value: 0.02199
> anova(lm.res)
Analysis of Variance Table
Response: values
Df Sum Sq Mean Sq F value Pr(>F)
group 2 416 208 4.16 0.02199 *
Residuals 45 2250 50
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
>
>
c/ms/2025/w07_anova_note.1744264836.txt.gz · Last modified: by hkimscil