Table of Contents
Type of Error
see hypothesis testing in r space
and hypothesis testing
Type I Error
###########################################
# type 1 error
###########################################
rm(list=ls())
rnorm2 <- function(n,mean,sd){
mean+sd*scale(rnorm(n))
}
# m.treated.s.but.not.work from p1
# = 104.022, red dot line
# type 1 error
set.seed(1292)
n.p <- 10000
m.p <- 100
sd.p <- 10
p1 <- rnorm2(n.p, m.p, sd.p)
m.p1 <- mean(p1)
sd.p1 <- sd(p1)
p2 <- rnorm2(n.p, m.p+5, sd.p)
m.p2 <- mean(p2)
sd.p2 <- sd(p2)
n.s <- 40
se.z1 <- c(sqrt(var(p1)/n.s))
se.z2 <- c(sqrt(var(p2)/n.s))
x.p1 <- seq(mean(p1)-5*se.z1,
mean(p2)+5*se.z1,
length.out = 500)
x.p2 <- seq(mean(p2)-5*se.z1,
mean(p2)+5*se.z1,
length.out = 500)
# Calculate the probability
# density for a normal distribution
y.p1 <- dnorm(x.p1, mean(p1), se.z1)
y.p2 <- dnorm(x.p2, mean(p2), se.z2)
# Plot the theoretical PDF
plot(x.p1, y.p1, type = "l",
lwd=3,
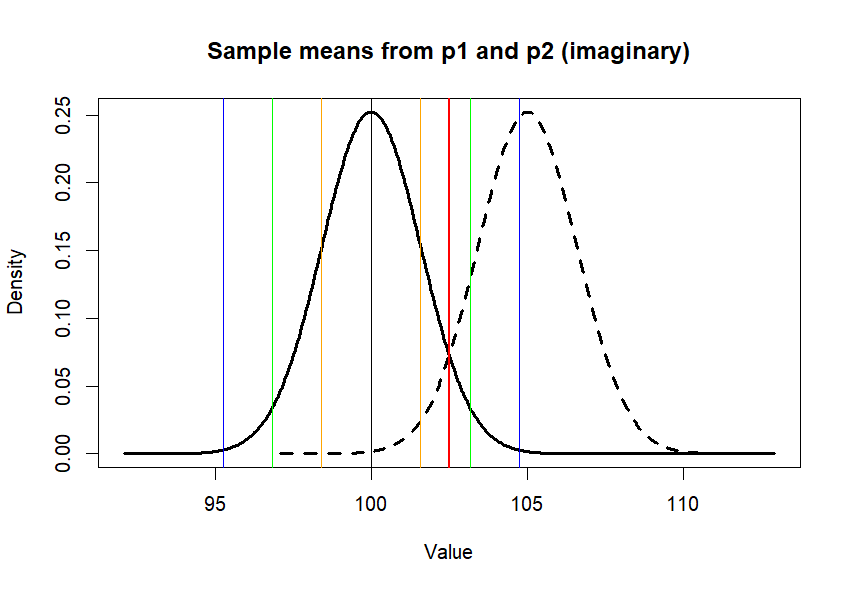
main = "Sample means from p1 and p2 (imaginary)",
xlab = "Value", ylab = "Density")
lines(x.p2, y.p2, lty=2, lwd=3)
m.p1 <- mean(p1)
se1 <- c(m.p1-se.z1, m.p1+se.z1)
se2 <- c(m.p1-2*se.z1, m.p1+2*se.z1)
se3 <- c(m.p1-3*se.z1, m.p1+3*se.z1)
abline(v=c(m.p1,se1,se2,se3),
col=c('black', 'orange', 'orange',
'green', 'green',
'blue', 'blue'),
lwd=1)
treated.s.but.not.work <- sample(p1, n.s)
m.treated.s.but.not.work <- mean(treated.s.but.not.work)
m.treated.s.but.not.work
# m.treated.s <- 103.1605 # set.seed(101)에서 얻은 treated.s 점수를 유지
abline(v=m.treated.s.but.not.work, col='red', lty=2, lwd=3)
se.z1
diff <- m.treated.s.but.not.work-mean(p1)
diff/se.z1
# usual way - using sample's variance
# instead of p1's variance to get
# standard error value
se.s <- sqrt(var(treated.s.but.not.work)/n.s)
se.s
diff/se.s
pt(diff/se.s, df=n.s-1,
lower.tail = F) * 2
t.test(treated.s.but.not.work, mu=m.p1, var.equal = T)
output
1
> ###########################################
> # type 1 error
> ###########################################
> rm(list=ls())
>
> rnorm2 <- function(n,mean,sd){
+ mean+sd*scale(rnorm(n))
+ }
>
> # m.treated.s.but.not.work from p1
> # = 104.022, red dot line
> # type 1 error
> set.seed(1292)
>
> n.p <- 10000
> m.p <- 100
> sd.p <- 10
>
> p1 <- rnorm2(n.p, m.p, sd.p)
> m.p1 <- mean(p1)
> sd.p1 <- sd(p1)
>
> p2 <- rnorm2(n.p, m.p+5, sd.p)
> m.p2 <- mean(p2)
> sd.p2 <- sd(p2)
>
…………………….
2
> n.s <- 40
> se.z1 <- c(sqrt(var(p1)/n.s))
> se.z2 <- c(sqrt(var(p2)/n.s))
>
> x.p1 <- seq(mean(p1)-5*se.z1,
+ mean(p2)+5*se.z1,
+ length.out = 500)
> x.p2 <- seq(mean(p2)-5*se.z1,
+ mean(p2)+5*se.z1,
+ length.out = 500)
>
> # Calculate the probability
> # density for a normal distribution
> y.p1 <- dnorm(x.p1, mean(p1), se.z1)
> y.p2 <- dnorm(x.p2, mean(p2), se.z2)
>
> # Plot the theoretical PDF
> plot(x.p1, y.p1, type = "l",
+ lwd=3,
+ main = "Sample means from p1 and p2 (imaginary)",
+ xlab = "Value", ylab = "Density")
> lines(x.p2, y.p2, lty=2, lwd=3)
>
>
> m.p1 <- mean(p1)
> se1 <- c(m.p1-se.z1, m.p1+se.z1)
> se2 <- c(m.p1-2*se.z1, m.p1+2*se.z1)
> se3 <- c(m.p1-3*se.z1, m.p1+3*se.z1)
> abline(v=c(m.p1,se1,se2,se3),
+ col=c('black', 'orange', 'orange',
+ 'green', 'green',
+ 'blue', 'blue'),
+ lwd=1)
>
> treated.s.but.not.work <- sample(p1, n.s)
> m.treated.s.but.not.work <- mean(treated.s.but.not.work)
> m.treated.s.but.not.work
[1] 104.022
> # m.treated.s <- 103.1605 # set.seed(101)에서 얻은 treated.s 점수를 유지
> abline(v=m.treated.s.but.not.work, col='red', lty=2, lwd=3)
>
…………………….
검은색 분포곡선 실선 (distribution of means from p1)
검은색 선 (mean of p1, 100)
노란색 (+- se)
연두색 (+- 2se)
파란색 (+- 3se)
검은색 분포곡선 점선 (distribution of means from p2)
붉은 색 점선 (A SAMPLE from p1 = 약의 효과가 없는 집단에서 (모집단에서) 추출한 sample)
그러나 이 위치에서 연구자는 약의 효과가 있다고 판단하게 된다.
왜냐하면 샘플의 평균이 녹색 선 밖에서 (.05 level보다 작은 위치에서) 나왔기 때문이다.
그러나, 현실로 보면 이 샘플은 p1에서 추출되었으므로 약의 효과가 없다고 판단해야 옳은 판단.
연구자는 판단에러를 범했다. 이것이 type 1 error.
검은색 점선 (distribution of means from p2, 평균 = 105): 약을 먹고 머리가 좋아진 모집단. 105로 5만큼 좋아졌다고 가정한 것. 사실, 머리가 좋아지는 약을 개발했다면 그 약을 먹은 모집단의 파라미터는 알수가 없다. 단지 105라고 가정한 것이다.
3
> se.z1 [1] 1.581139 > > diff <- m.treated.s.but.not.work-mean(p1) > diff/se.z1 [1] 2.543733 > > # usual way - using sample's variance > # instead of p1's variance to get > # standard error value > se.s <- sqrt(var(treated.s.but.not.work)/n.s) > se.s [1] 1.851968 > diff/se.s [1] 2.171741 > > pt(diff/se.s, df=n.s-1, + lower.tail = F) * 2 [1] 0.03601933 > t.test(treated.s.but.not.work, mu=m.p1, var.equal = T) One Sample t-test data: treated.s.but.not.work t = 2.1717, df = 39, p-value = 0.03602 alternative hypothesis: true mean is not equal to 100 95 percent confidence interval: 100.276 107.768 sample estimates: mean of x 104.022 >
…………………….
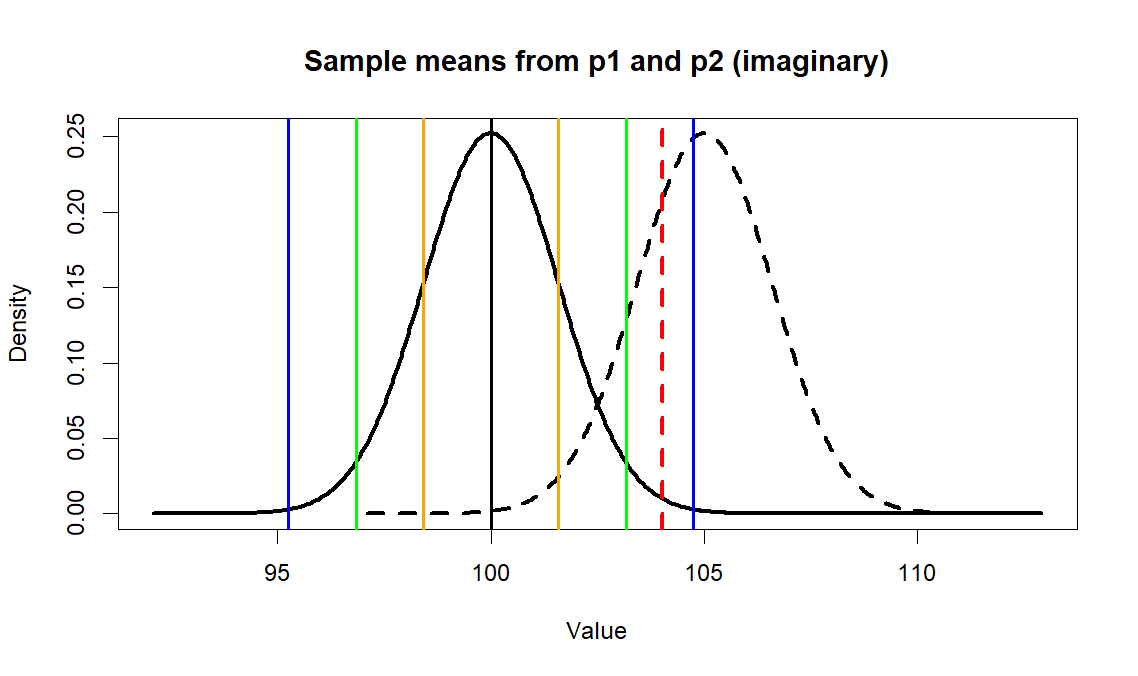
그러나, 이 샘플의 평균이 p1에 속한다고 하고, 이 평균값이 나올 확률을 알아보려면
pnorm(m.treated.s, mean=m.p1, sd=se.z, lower.tail=F) * 2
일 것이다. 그리고,
이 빨간 선에 해당하는 점수 m.treated.s의 표준점수는 (이 점수가 p1에 속한다고 가정했을 때)
\begin{eqnarray*}
& & \dfrac{(\text{m.treated.s} - \text{m.p1})} {se.z} \\
& & = \dfrac{\text{diff}} {\text{se.z}} \\
& & = 1.572729
\end{eqnarray*}
우리는 이 점수가 +- 2 점을 넘는가를 보려고 한것이다. 그러나, 2점을 넘지 못하기에 이 점수가 p1에서 나올만 한 점수라고 인정할 수 밖에 없다. 즉, 영가설을 부정하려고 한것이 실패하여, 원래의 연구가설을 검증을 하지 못하게 된다. 즉, 약의 효과가 있다고 주장할 수 없게 된다. 그러나, 우리가 m.treated.s 값을 얻은 경로를 보면 이 값은 분명 p2에서 나왔으므로 연구가설 검증에 성공했어야 했다. 따라서 우리가 내린 판단은 잘못된 것이라고 하겠다. 이런 잘못된 판단을 Type I Error라고 한다.
cm.treated.s = 102.4867 -- > RED LINE
This red line came from p2, whose mean is different from p1's. We know that p2's mean is 5 greater than p1's (100). And red line (mean of a sample from p2, whose sample size (s.size) 40. So the truth is that we should deny null hypothesis, and accept the alternative (research) one. But, because of the value, m.treated.s we could not. We failed to find out the truth (ERROR). This kind of error is called Type I Error.