c:ms:2025:lecture_note_week_04
Table of Contents
Variance
Why n-1 for 모집단
# variance # why we use n-1 when calculating # population variance (instead of N) a <- rnorm2(100000000, 100, 10) a.mean <- mean(a) ss <- sum((a-a.mean)^2) n <- length(a) df <- n-1 ss/n ss/df
Variance output
> # variance > # why we use n-1 when calculating > # population variance (instead of N) > a <- rnorm2(100000000, 100, 10) > a.mean <- mean(a) > ss <- sum((a-a.mean)^2) > n <- length(a) > df <- n-1 > ss/n [1] 100 > ss/df [1] 100 >
rule of SD
# standard deviation 68, 85, 99 rule one.sd <- .68 two.sd <- .95 thr.sd <- .99 1-one.sd (1-one.sd)/2 qnorm(0.16) qnorm(1-0.16) 1-two.sd (1-two.sd)/2 qnorm(0.025) qnorm(0.975) 1-thr.sd (1-thr.sd)/2 qnorm(0.005) qnorm(1-0.005)
rule of SD output
> # standard deviation 68, 85, 99 rule > one.sd <- .68 > two.sd <- .95 > thr.sd <- .99 > 1-one.sd [1] 0.32 > (1-one.sd)/2 [1] 0.16 > qnorm(0.16) [1] -0.9944579 > qnorm(1-0.16) [1] 0.9944579 > > 1-two.sd [1] 0.05 > (1-two.sd)/2 [1] 0.025 > qnorm(0.025) [1] -1.959964 > qnorm(0.975) [1] 1.959964 > > 1-thr.sd [1] 0.01 > (1-thr.sd)/2 [1] 0.005 > qnorm(0.005) [1] -2.575829 > qnorm(1-0.005) [1] 2.575829 > >
Sampling distribution
OR the distribution of sample means
# sampling distribution
n.ajstu <- 100000
mean.ajstu <- 100
sd.ajstu <- 10
set.seed(1024)
ajstu <- rnorm2(n.ajstu, mean=mean.ajstu, sd=sd.ajstu)
mean(ajstu)
sd(ajstu)
var(ajstu)
min.value <- min(ajstu)
max.value <- max(ajstu)
min.value
max.value
iter <- 10000 # # of sampling
n.4 <- 4
means4 <- rep (NA, iter)
for(i in 1:iter){
means4[i] = mean(sample(ajstu, n.4))
}
n.25 <- 25
means25 <- rep (NA, iter)
for(i in 1:iter){
means25[i] = mean(sample(ajstu, n.25))
}
n.100 <- 100
means100 <- rep (NA, iter)
for(i in 1:iter){
means100[i] = mean(sample(ajstu, n.100))
}
n.400 <- 400
means400 <- rep (NA, iter)
for(i in 1:iter){
means400[i] = mean(sample(ajstu, n.400))
}
n.900 <- 900
means900 <- rep (NA, iter)
for(i in 1:iter){
means900[i] = mean(sample(ajstu, n.900))
}
n.1600 <- 1600
means1600 <- rep (NA, iter)
for(i in 1:iter){
means1600[i] = mean(sample(ajstu, n.1600))
}
n.2500 <- 2500
means2500 <- rep (NA, iter)
for(i in 1:iter){
means2500[i] = mean(sample(ajstu, n.2500))
}
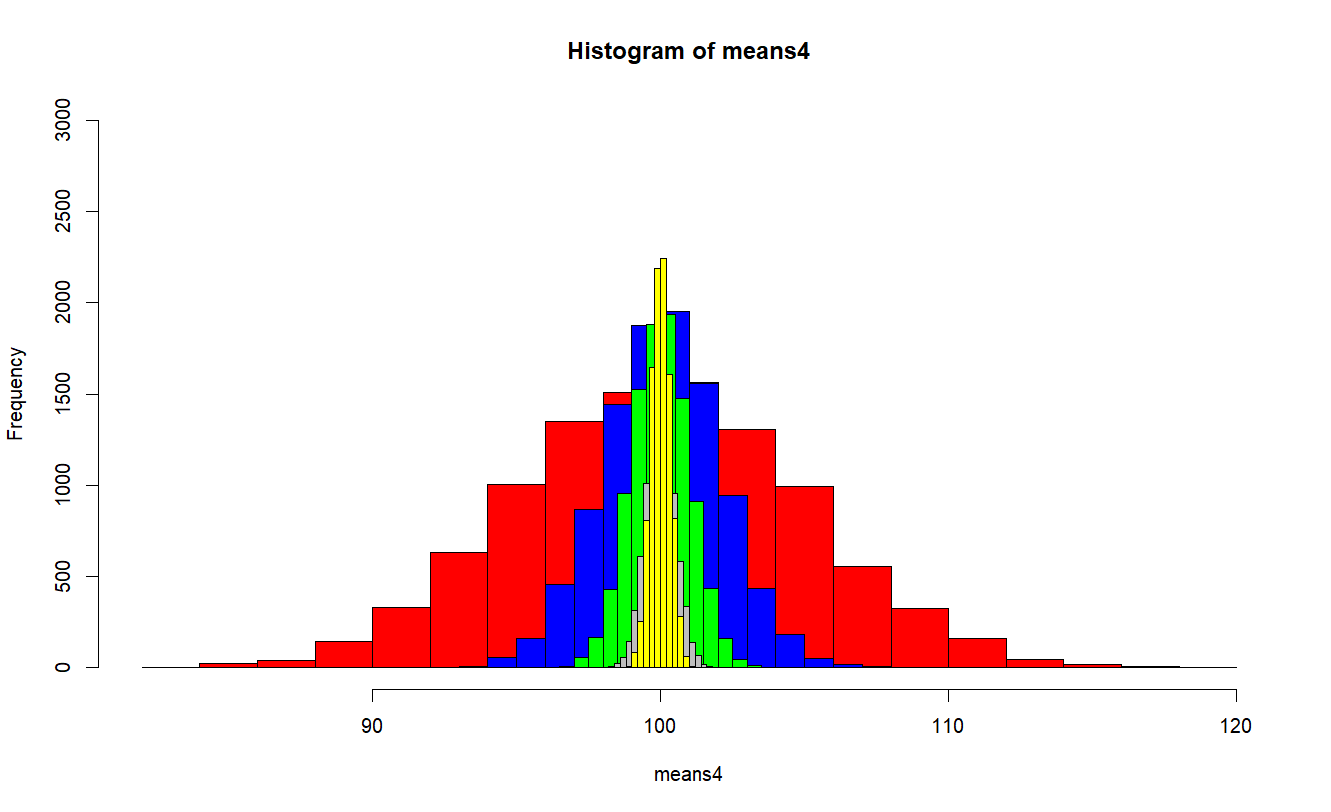
h4 <- hist(means4)
h25 <- hist(means25)
h100 <- hist(means100)
h400 <- hist(means400)
h900 <- hist(means900)
h1600 <- hist(means1600)
h2500 <- hist(means2500)
plot(h4, ylim=c(0,3000), col="red")
plot(h25, add = T, col="blue")
plot(h100, add = T, col="green")
plot(h400, add = T, col="grey")
plot(h900, add = T, col="yellow")
sss <- c(4,25,100,400,900,1600,2500) # sss sample sizes
ses <- rep (NA, length(sss)) # std errors
for(i in 1:length(sss)){
ses[i] = sqrt(var(ajstu)/sss[i])
}
ses.means4 <- sqrt(var(means4))
ses.means25 <- sqrt(var(means25))
ses.means100 <- sqrt(var(means100))
ses.means400 <- sqrt(var(means400))
ses.means900 <- sqrt(var(means900))
ses.means1600 <- sqrt(var(means1600))
ses.means2500 <- sqrt(var(means2500))
ses.real <- c(ses.means4, ses.means25,
ses.means100, ses.means400,
ses.means900, ses.means1600,
ses.means2500)
ses.real
ses
se.1 <- ses
se.2 <- 2 * ses
lower.s2 <- mean(ajstu)-se.2
upper.s2 <- mean(ajstu)+se.2
data.frame(cbind(sss, ses, ses.real, lower.s2, upper.s2))
min.value <- min(ajstu)
max.value <- max(ajstu)
min.value
max.value
# means25 분포에서 population의 가장 최소값인
# min.value가 나올 확률은 얼마나 될까?
m.25 <- mean(means25)
sd.25 <- sd(means25)
m.25
sd.25
pnorm(min.value, m.25, sd.25)
# 위처럼 최소값, 최대값의 probability를 가지고
# 확률을 구하는 것은 의미가 없음. 즉, 어떤 샘플의
# 평균이 원래 population에서 나왔는지를 따지는 것을
# 그 pop의 최소값으로 판단하는 것은 의미가 없음.
# 그 보다는 sd.25와 같은 standard deviation을 가지
# 고 보는 것이 타당함. 이 standard deviation 을
# standard error라고 부름
se.25 <- sd.25
se.25.2 <- 2 * sd.25
mean(means25)+c(-se.25.2, se.25.2)
# 위처럼 95% certainty를 가지고 구간을 판단하거나
# 아래 처럼 점수를 가지고 probability를 볼 수도
# 있음
pnorm(95.5, m.25, sd.25)
# 혹은
pnorm(95.5, m.25, sd.25) * 2
# 매우중요. 위의 이야기는 내가 25명의 샘플을 취
# 했을 때, 그 점수가 95.5가 나올 확률을 구해 본것
# 다른 예로
pnorm(106, m.25, sd.25, lower.tail = F)
# 혹은
2 * pnorm(106, m.25, sd.25, lower.tail = F)
Sampling distribution output
> # sampling distribution
> n.ajstu <- 100000
> mean.ajstu <- 100
> sd.ajstu <- 10
>
> set.seed(1024)
> ajstu <- rnorm2(n.ajstu, mean=mean.ajstu, sd=sd.ajstu)
>
> mean(ajstu)
[1] 100
> sd(ajstu)
[1] 10
> var(ajstu)
[,1]
[1,] 100
> min.value <- min(ajstu)
> max.value <- max(ajstu)
> min.value
[1] 57.10319
> max.value
[1] 141.9732
>
> iter <- 10000 # # of sampling
>
> n.4 <- 4
> means4 <- rep (NA, iter)
> for(i in 1:iter){
+ means4[i] = mean(sample(ajstu, n.4))
+ }
>
> n.25 <- 25
> means25 <- rep (NA, iter)
> for(i in 1:iter){
+ means25[i] = mean(sample(ajstu, n.25))
+ }
>
> n.100 <- 100
> means100 <- rep (NA, iter)
> for(i in 1:iter){
+ means100[i] = mean(sample(ajstu, n.100))
+ }
>
> n.400 <- 400
> means400 <- rep (NA, iter)
> for(i in 1:iter){
+ means400[i] = mean(sample(ajstu, n.400))
+ }
>
> n.900 <- 900
> means900 <- rep (NA, iter)
> for(i in 1:iter){
+ means900[i] = mean(sample(ajstu, n.900))
+ }
>
> n.1600 <- 1600
> means1600 <- rep (NA, iter)
> for(i in 1:iter){
+ means1600[i] = mean(sample(ajstu, n.1600))
+ }
>
> n.2500 <- 2500
> means2500 <- rep (NA, iter)
> for(i in 1:iter){
+ means2500[i] = mean(sample(ajstu, n.2500))
+ }
>
> h4 <- hist(means4)
> h25 <- hist(means25)
> h100 <- hist(means100)
> h400 <- hist(means400)
> h900 <- hist(means900)
> h1600 <- hist(means1600)
> h2500 <- hist(means2500)
>
>
> plot(h4, ylim=c(0,3000), col="red")
> plot(h25, add = T, col="blue")
> plot(h100, add = T, col="green")
> plot(h400, add = T, col="grey")
> plot(h900, add = T, col="yellow")
>
>
> sss <- c(4,25,100,400,900,1600,2500) # sss sample sizes
> ses <- rep (NA, length(sss)) # std errors
> for(i in 1:length(sss)){
+ ses[i] = sqrt(var(ajstu)/sss[i])
+ }
> ses.means4 <- sqrt(var(means4))
> ses.means25 <- sqrt(var(means25))
> ses.means100 <- sqrt(var(means100))
> ses.means400 <- sqrt(var(means400))
> ses.means900 <- sqrt(var(means900))
> ses.means1600 <- sqrt(var(means1600))
> ses.means2500 <- sqrt(var(means2500))
> ses.real <- c(ses.means4, ses.means25,
+ ses.means100, ses.means400,
+ ses.means900, ses.means1600,
+ ses.means2500)
> ses.real
[1] 4.9719142 2.0155741 0.9999527 0.5034433 0.3324414
[6] 0.2466634 0.1965940
>
> ses
[1] 5.0000000 2.0000000 1.0000000 0.5000000 0.3333333
[6] 0.2500000 0.2000000
> se.1 <- ses
> se.2 <- 2 * ses
>
> lower.s2 <- mean(ajstu)-se.2
> upper.s2 <- mean(ajstu)+se.2
> data.frame(cbind(sss, ses, ses.real, lower.s2, upper.s2))
sss ses ses.real lower.s2 upper.s2
1 4 5.0000000 4.9719142 90.00000 110.0000
2 25 2.0000000 2.0155741 96.00000 104.0000
3 100 1.0000000 0.9999527 98.00000 102.0000
4 400 0.5000000 0.5034433 99.00000 101.0000
5 900 0.3333333 0.3324414 99.33333 100.6667
6 1600 0.2500000 0.2466634 99.50000 100.5000
7 2500 0.2000000 0.1965940 99.60000 100.4000
>
> min.value <- min(ajstu)
> max.value <- max(ajstu)
> min.value
[1] 57.10319
> max.value
[1] 141.9732
> # means25 분포에서 population의 가장 최소값인
> # min.value가 나올 확률은 얼마나 될까?
> m.25 <- mean(means25)
> sd.25 <- sd(means25)
> m.25
[1] 100.0394
> sd.25
[1] 2.015574
> pnorm(min.value, m.25, sd.25)
[1] 5.414963e-101
> # 위처럼 최소값, 최대값의 probability를 가지고
> # 확률을 구하는 것은 의미가 없음. 즉, 어떤 샘플의
> # 평균이 원래 population에서 나왔는지를 따지는 것을
> # 그 pop의 최소값으로 판단하는 것은 의미가 없음.
> # 그 보다는 sd.25와 같은 standard deviation을 가지
> # 고 보는 것이 타당함. 이 standard deviation 을
> # standard error라고 부름
> se.25 <- sd.25
> se.25.2 <- 2 * sd.25
> mean(means25)+c(-se.25.2, se.25.2)
[1] 96.00822 104.07051
> # 위처럼 95% certainty를 가지고 구간을 판단하거나
> # 아래 처럼 점수를 가지고 probability를 볼 수도
> # 있음
> pnorm(95.5, m.25, sd.25)
[1] 0.01215658
> # 혹은
> pnorm(95.5, m.25, sd.25) * 2
[1] 0.02431317
> # 매우중요. 위의 이야기는 내가 25명의 샘플을 취
> # 했을 때, 그 점수가 95.5가 나올 확률을 구해 본것
> # 다른 예로
> pnorm(106, m.25, sd.25, lower.tail = F)
[1] 0.001551781
> # 혹은
> 2 * pnorm(106, m.25, sd.25, lower.tail = F)
[1] 0.003103562
>
>
>
mean and variance of sample means
위에서 standard deviation of sample means (샘플평균들로 이루어진 집합의 표준편차) 값을 standard error라고 부른다. 그리고 이 standard error 값은 원래 population의 $\sigma^2$ 값을 샘플의 사이즈로 나누어준 값을 ($\sigma^2/n$) 갖게 된다. 또한 이 샘플평균들의 집합의 평균은 (샘플평균들의 기대값, the mean of sample means) population의 평균값을 ($\mu$) 갖게 된다. 이에 대한 수학적 증명 문서는 mean and variance of the sample mean.
Hypothesis testing
먼저 이해하기
- pnorm (and rnorm, qnorm)
- 표준점수
- sampling distribution
- = distribution of sample means
- = distribution of the sample mean
- standard error
- = standard deviation of sample means
- = standard deviation of sampling distribution
- CLT
- $\overline{X} \sim N(\mu, \displaystyle \frac{\sigma^2}{n}) $
위를 이해한다면 이제 우리는 차이의가설을 만들어 테스트해볼 수 있다. hypothesis testing
# 가설 테스트
set.seed(2000)
pop <- rnorm2(1000000, 50, 10)
m.pop <- mean(pop)
sd.pop <- sd(pop)
var.pop <- var(pop)
# 시뮬레이션 시작
iter <- 10000 # 사실은 무한대
n <-100 # sample size
means <-rep(NA, iter) # means memory reservation
for(i in 1:iter){
means[i] = mean(sample(pop, n))
}
m.empirical <- mean(means)
se.empirical <- sd(means)
m.empirical
se.empirical
se <- sd.pop/sqrt(n)
se2 <- se * 2
se3 <- se * 3
vlines <- c(m.empirical,
m.empirical+c(-se,se),
m.empirical+c(-se2,se2),
m.empirical+c(-se3,se3))
colors = c("blue",
"red", "red",
"darkgreen", "darkgreen",
"black", "black")
hist(means)
abline(v=vlines, col=colors, lwd=2, lty="dashed")
# 아래는 n=100 샘플을 하여 평균을 구해본 것
s1 <- sample(pop, n)
s1
mean(s1)
s2 <- sample(pop, n)
mean(s2)
# set.seed(2000)
iter <- 20
n <-100
means <-rep(NA, iter)
for(i in 1:iter){
means[i] = mean(sample(pop, n))
}
means
means < 48
means > 52
table(means<48)
table(means>52)
table(means<48|means>52)
s3 <- rnorm2(n, 53.2, 10)
m.s3 <- mean(s3)
m.s3
p.val.one.tail <-
pnorm(m.s3, m.pop, se, lower.tail = F)
p.val.two.tail <-
pnorm(m.s3, m.pop, se, lower.tail = F) * 2
p.val.one.tail
p.val.two.tail
set.seed(1010)
s4 <- rnorm(100, 54, 10)
m.s4 <- mean(s4)
m.s4
p.val.two.tail <- pnorm(m.s4,
m.pop,
se, lower.tail = F) * 2
p.val.two.tail
p.val.one.tail <-p.val.two.tail/2
p.val.one.tail
z.score <- (m.s4-m.pop)/se
z.score
pnorm(z.score, lower.tail = F)*2
# 만약에 pnorm을 이용하여 정확한
# probability를 구할 수 없는 상황
# 이라고 가정하면
# 우리는 CLT 정리를 생각하여
# n = 100인 샘플을 무한 반복하여
# 샘플링 하고, 각 샘플의 평균을
# 기록하여 모아둔다면 그 분포는
# N(mu, sigma^2/n) 이라는 것을
# 안다. N(50, 100/100)
# 따라서 이 집합의 표준편차값은
# sigma/sqrt(n) = 1
# 대충 바로 위의 그래프와 같은
# 모습이고, 이 그래프에서 sd를
# 위로 두개, 밑으로 두개 쓴 구간은
# 녹색에 해당하는 48-52 이다.
# 이 구간에서 샘플의 평균이 나타날
# 확률은 95%라고 하였다. 따라서
# 우리가 가지고 있는 m.s4의 값은
# 52점 밖에 존재하므로
# 5%의 확률로서만 얻을 수 있는 샘플
# 평균이라는 것을 알수 있다.
# 위의 결과를 가지고 두가지 판단을 할 수
# 있다. 하나는 이 샘플의 평균이 모집단
# 평균이 50이고 표준편차가 10인 집단에서
# 나올 수 있는 샘플일 확률이 1/20밖에
# 안되므로 이 샘플은 우리가 염두에 둔
# 원래 모집단에서 나온 것이 아니라는
# 생각이다.
# 두 번째는 이 샘플이 그 모집단에서
# 나오기는 했다. 그러나 1/20 확률에
# 우연찮게 이번에 걸려서 m.s4의 평균값이
# 나왔다.
# 이것은 1/20확률에 근거한 판단이므로 첫
# 번째 판단을 나의 주 판단으로 한다. 즉,
# 이 샘플은 원래 모집단에서 나올 샘플이
# 아닌 다른 집단에서 나온 샘플이다.
# 그런데 여기서 추가로 주목할 점은 내가 샘플이
# 정상적인지 아니면 비정상적인지 판단하는 기준
# 선을 표준점수 +- 2에 두었다는 점이다.
# m.s4의 점수는 53.2 이므로 m.s4-m.pop(50)
# 인 3.2는 샘플평균들의 집합에서 구하는
# 표준편차가 3.2만큼 떨어진 것을 알 수 있다.
# 우리는 이것을 표준점수라고 하였으므로
# 우리가 구한 z-score = 3.2라는 것을 알 수 있고
# 이 점수가 표준편차값 2개를 위아래로 적용한 선보다
# 밖에 존재하므로 샘플이 모집단에서 나오지 않은
# 특별한 샘플이라는 것을 주장할 수 있다.
c/ms/2025/lecture_note_week_04.txt · Last modified: by hkimscil